检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
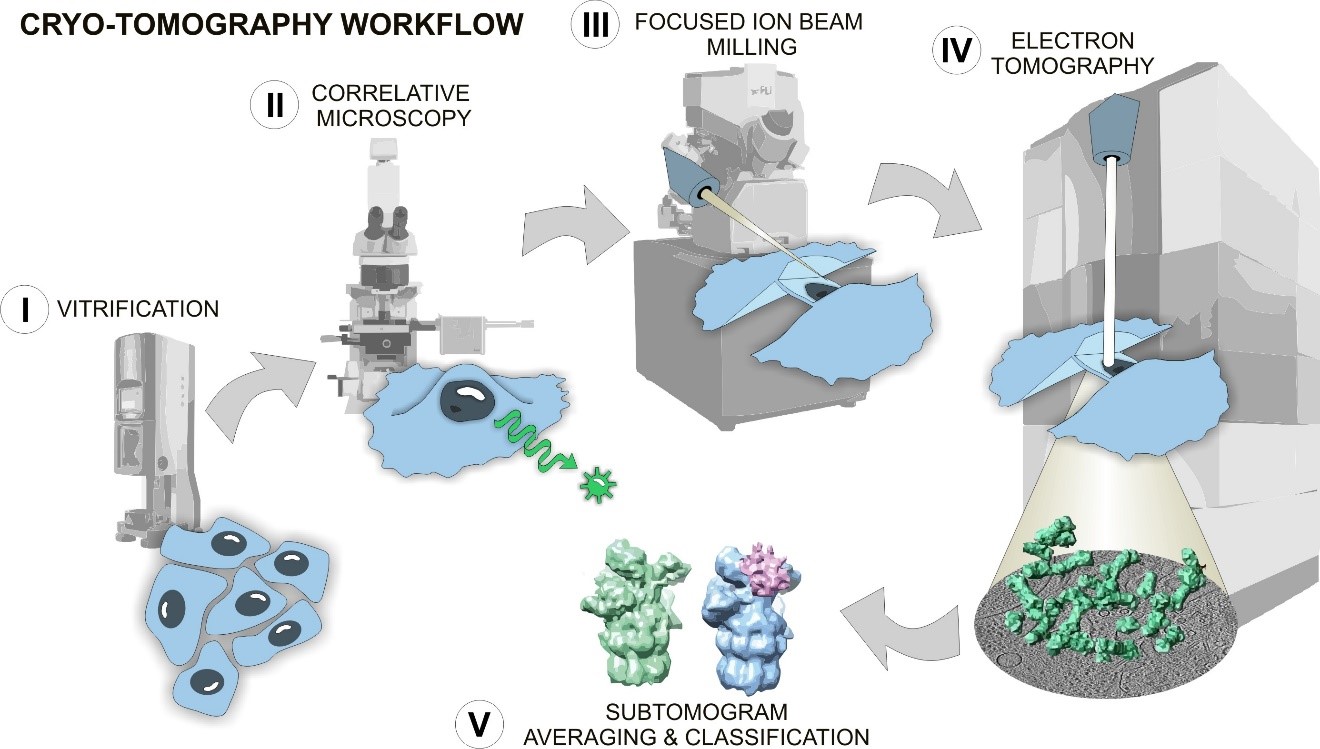
本实验室依托于威尼斯wnsr666和北大-清华生命科学联合中心,主要运用冷冻光电联用(CLEM)、冷冻电子断层扫描(cryo-ET)、冷冻电镜(cryo-EM)等多尺度成像技术对疾病相关细胞生物学问题进行研究。现因工作需要,公开招聘博士后研究人员。
【岗位职责】
1.能够独立承担实验室的科研任务,推动研究项目的开展;
2.协助PI指导博士生和技术员;
【应聘要求】
1. 已获得或即将获得博士学位;
2. 结构生物学、细胞生物学背景,也欢迎其他学科背景且熟练掌握cryo-TEM或FIB/SEM技能的申请者;
3. 熟悉matlab或python使用者优先;
4. 工作严谨认真,条理清晰,积极主动,有责任心;
【待遇条件】
在威尼斯wnsr666博士后相关规定基础上提供有市场竞争性待遇条件,并可以协助申请北大博雅博士后计划以及北大-清华生命科学联合中心博士后基金等资助;
【招聘方式】
请将申请信、个人简历以及2-3名推荐人信息发送到:qguo2020@pku.edu.cn。邮件标题请注明“应聘-博士后申请-姓名”。我们将根据材料对所有应聘者进行初选,通过初选者将邮件通知面试并择优录用。
本招聘启事自发布之日起,到所需岗位招满为止。感谢关注,并期待您的加入!
PI履历:

2020.08起
威尼斯wnsr666助理教授(Tenure-Track),北大-清华生命科学联合中心研究员
2014.9-2020.07
德国马普生化所(Max-Planck Institute of Biochemistry)博士后研究员,洪堡学者,EMBO long-term fellowship
2009.09-2014.06
清华大学威尼斯wnsr666博士
代表论文:
Guo, Q., Lehmer, C., Martinez-Sanchez, A., Rudack, T., Beck, F., Hartmann, H., Hipp, M.S., Hartl, F.U., Edbauer, D., Baumeister, W., Fernandez-Busnadiego, R. (2018) In Situ Structure of Neuronal C9orf72 Poly-GA Aggregates Reveals Proteasome Recruitment.Cell172, 696-705.e612.Bioart推送
Guo, Q., Bin, H., Cheng, J., Seefelder, M., Engler, T., Pfeifer, G., Oeckl, P., Otto, M., Moser, F., Maurer, M., Pautsch, A., Baumeister, W., Fernandez-Busnadiego, R., Kochanek, S. (2018). The cryo-electron microscopy structure of huntingtin.Nature555, 117–120.
Yasuda, S., Tsuchiya, H., Kaiho, A.,Guo, Q., Ikeuchi, K., Endo, A., Arai, N., Ohtake, F., Murata, S., Inada, T., et al. (2020). Stress- and ubiquitylation-dependent phase separation of the proteasome.Nature578, 296–300.
Rigort, A.,Guo, Q., Bäuerlein, F.J.B., and Fernández-Busnadiego, R. (2018). The combination of cryo-FIB and in situ cryo-electron tomography enables ultrastructural analysis of disease-related protein aggregates.Microsc. Anal. S4. (封面文章)
Zhao, Y., Zeng, X.,Guo, Q., and Xu, M. (2018). An integration of fast alignment and maximum-likelihood methods for electron subtomogram averaging and classification.Bioinformatics34, i227–i236