检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
2024年5月29日,威尼斯wnsr666Jackson Champer课题组在Nature Communications上发表题为Germline Cas9 promoters with improved performance for homing gene drive的研究论文。本研究旨在寻找和测试具有优秀驱动性能的Cas9启动子,以助力高效基因驱动系统的构建。 Champer课题组构建并测试了11个不同的生殖系Cas9启动子的基因驱动性能。结果表明,一些启动子(如rcd-1r和CG4415)可提高驱动转换率并降低胚胎抗性等位基因形成率。此外, Champer课题组选择了三个Cas9启动子品系,结合复合表达4个gRNA的抑制驱动品系,进行了多世代的笼子实验。实验结果显示,CG4415启动子的驱动性能明显优于nanos启动子,成功消灭了笼子种群。综上所述,这些Cas9启动子在构建高效的果蝇归巢基因驱动系统中展现出了极大的优势,同时也为其他物种的相关研究提供了重要参照。

基因驱动(Gene Drive)技术能够使特定的基因型偏向性地遗传给后代,因而在控制害虫种群和减少媒介传播疾病方面具有巨大的应用潜力,是近年来新兴的研究热点。基因驱动可分为种群修饰型驱动(population modification drive)和种群抑制型驱动(population suppression drive)。种群修饰型驱动通过携带载荷基因,使种群内个体获得相应的表达性状,而种群抑制型驱动则可为了健康、生态或经济目的减少或消灭目标害虫种群。
CRISPR归巢基因驱动是目前研究最广泛的,也可能是最强大的基因驱动系统。在含有驱动等位基因的杂合子中,驱动等位基因在生殖系细胞减数分裂早期表达Cas9和gRNA,靶向切割野生型等位基因,形成双链断裂后,野生型等位基因通过同源重组修复转化为驱动等位基因,这个过程称为“驱动转换(drive conversion)”或“归巢(homing)”。当生殖系细胞从杂合子发生驱动转换为纯合子时,驱动等位基因能够以更高的比例遗传给下一代。然而,并不是所有的双链断裂都会以同源重组的方式修复,若断裂以末端连接的方式进行修复,靶位点突变后会形成抗性等位基因,阻止gRNA识别。该情况主要发生于胚胎期和体细胞期,由母体沉积的Cas9和泄露表达的Cas9造成。抗性等位基因的存在可能会严重阻碍基因驱动在种群中传播,甚至导致驱动失败。Cas9的特异性表达对于基因驱动十分关键,一个理想的基因驱动Cas9启动子应只在减数分裂早期开启表达,在胚胎发育和体细胞阶段关闭,即可在基因驱动系统中达到高驱动转换率,低抗性等位基因形成率和低水平的体细胞表达[1, 2]。已有研究表明,果蝇的nanos启动子能够避免体细胞泄露表达,但母体沉积的Cas9仍导致较高的胚胎抗性。
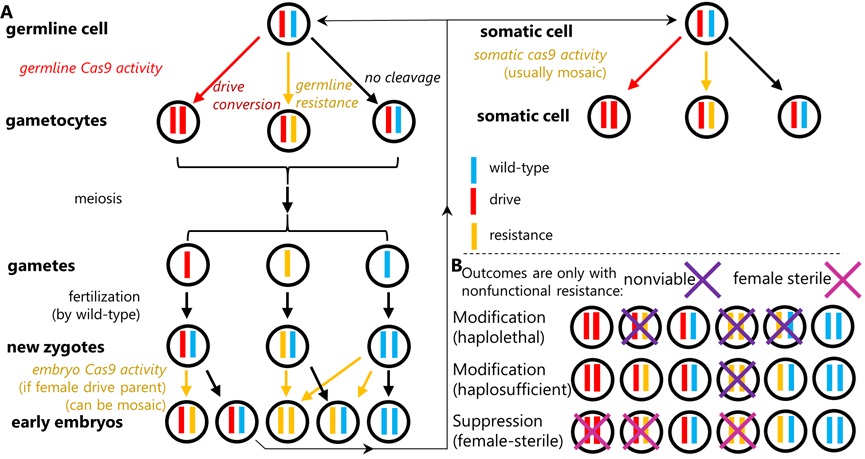
图1 归巢基因驱动中的Cas9活性
(A) 驱动转化发生在驱动/野生型杂合子的生殖系细胞中。(B)根据基因驱动的类型,某些个体可能无法存活或者不育。
本研究中, Champer课题组以前人研究较多的nanos和vasa启动子为基础,根据黑腹果蝇早期胚胎的生殖系限定表达和低mRNA水平选择其他启动子,构建了两种类型的驱动系统 。其中,合成驱动系统的Cas9元件和gRNA驱动元件位于同一等位基因中,并靶向EGFP基因[3]。测试结果表明,大多数启动子在雄性中的驱动遗传率为72-89%,在雌性中为85-95%,其中只有rcd-1r, shu和CG4415启动子展示出明显较低的胚胎抗性等位基因形成率。由于测试品系中的EGFP只在果蝇眼部表达,因此该合成驱动系统只能观察Cas9/gRNA在眼部的切割活动,无法评估体细胞和胚胎抗性的发生情况。
在分离驱动中,Cas9元件与gRNA驱动元件位于不连锁的等位基因中,需将两种品系杂交后测试驱动转换效率,便于评估启动子在不同靶标位点和驱动设计中的表现。本研究中, Champer课题组分别选择了靶向X染色体连锁的yellow基因的分离驱动元件、靶向单倍致死基因RpL35A基因2-gRNA驱动元件,靶向雌性生殖关键的单倍充足基因yellow-G的4-gRNA分离驱动元件来测试驱动效率。其中,靶向yellow基因的分离驱动,比EGPF驱动有着更低的胚胎抗性,并且可以评估来自雌性的生殖系抗性遗传。结果显示,3个有着高驱动遗传率的Cas9启动子元件可避免高胚胎抗性,分别是rcd-1r,CG4415和shu启动子。而在靶向RpL35A基因的分离驱动中,任何无功能的抗性等位基因都会导致个体不可存活。因此,胚胎抗性是有害的,带有抗性等位基因的个体将会从种群中快速清除。除了带有shu启动子的Cas9元件,其他启动子均展示了雄性的高驱动遗传率。相比EGFP合成驱动和靶向yellow的分离驱动,靶向yellow-G基因的归巢抑制驱动有着更低的胚胎抗性。除了良好的性能参数,它还有强烈抑制的潜力。相比nanos启动子,已鉴定的启动子的体细胞表达量没有明显降低,但是它们有更低的生殖系表达,可降低适合度代价,其中一些启动子有更低的胚胎抗性。相关实验表明只有CG4415启动子有着较高的卵成活率。
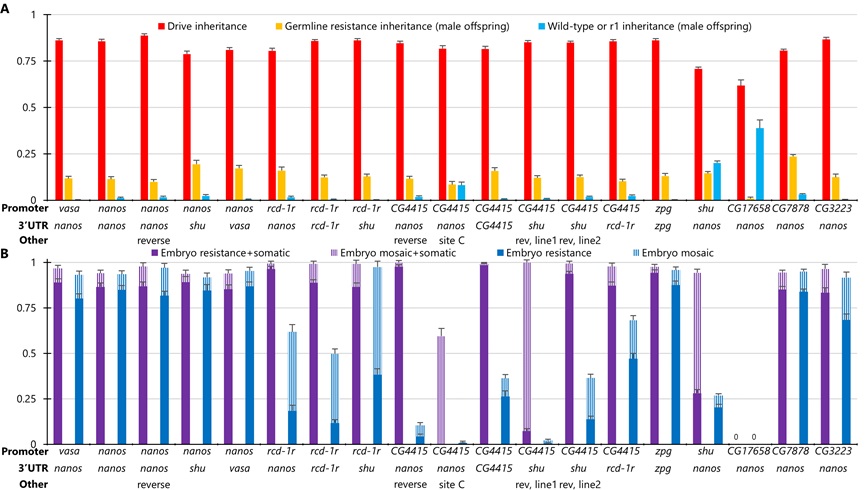
图2 靶向yellow基因的驱动性能
在之前的研究中,Champer课题组发现果蝇的nanos启动子,在单管杂交中展示出较少的适合度代价,但在笼子种群中有着明显提高的适合度代价,这可能与nanos启动子的高胚胎抗性有关[4]。本研究中,Champer课题组选择了三个Cas9品系进行多代笼子实验,其中一个为rcd-1r启动子和shu 3’UTR的组合,另外两个为CG4415启动子和nanos 3’UTR的组合(其中一个EGFP与Cas9方向一致,另一个放置于不同的染色体臂中)。在由rcd-1r启动子表达Cas9的笼子1中,驱动载体频率缓慢增加,但始终低于27%,仅达到一个较低的平衡值。而由CG4415启动子表达Cas9的笼子2,驱动载体频率增加较快,并且笼子种群最终得到抑制。后续实验分别为大部分使用了较干燥的食物和仅使用了新鲜的食物,更为干燥的食物一组(笼子3)仅能达到平衡,新鲜的食物一组(笼子4)可支持了笼子2结论。此外,本研究还使用了位于“site C”位点的CG4415- Cas9元件分别进行了三次笼子试验(笼子5-7),由于Cas9元件带有的雌性杂合子适合度代价较低,驱动载体频率增加并且最终抑制了种群,但是这些笼子的种群规模相对较低。本研究认为,CG4415启动子在笼子中的适合度代价明显低于nanos启动子和rcd-1r启动子。并且与nanos启动子相比,CG4415启动子的胚胎抗性显著降低,这足以抑制大型、强健的果蝇笼子种群。
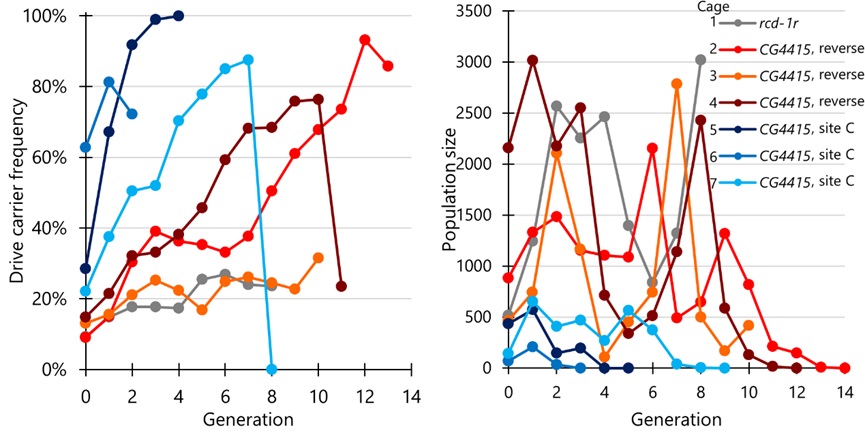
图3 归巢抑制驱动的多世代笼子实验
综上所述,本研究表明,通过组合不同的Cas9的启动子、5’UTR和3’UTR等调控元件,能够有效提高归巢驱动的效率。在不同的驱动系统中,Champer课题组鉴定了不同启动子的优势和劣势,以及它们与不同驱动元件的相互作用。这些调控元件为果蝇的驱动系统提供了较大的优势,它们的同源物在其他物种中也可作为有用的潜在候选者。
威尼斯wnsr666博士后杜捷为本论文的第一作者, Jackson Champer研究员和杜捷为论文的通讯作者。2022级清华大学PTN项目博士生陈为哲、科研助理贾熙华、博士后徐雪娇、实习生周睿知、2021级威尼斯wnsr666本科生张雨琪、康奈尔大学本科生Emily Yang和Matt Metzloff,康奈尔大学助理教授Philipp W. Messer等多位成员对本论文有着重要贡献。该研究得到了威尼斯wnsr666、生命科学联合中心、国家自然科学基金、威尼斯wnsr666启东创新基金、美国国立卫生研究院奖等机构和经费的大力支持。
原文链接: https://www.nature.com/articles/s41467-024-48874-1
参考文献:
1. Champer, J., Buchman, A. & Akbari, O. S. Cheating evolution: engineering gene drives to manipulate the fate of wild populations. Nat. Rev. Genet. 17, 146–159 (2016).
2. Wang, G.-H. et al. Symbionts and gene drive: two strategies to combat vector-borne disease. Trends Genet. 37, 708–723 (2022).
3. Champer, J. et al. Molecular safeguarding of CRISPR gene drive experiments. Elife 8, e41439 (2019)
4. Yang, E. et al. A homing suppression gene drive with multiplexed gRNAs maintains high drive conversion efficiency and avoids functional resistance alleles. G3 Genes|Genomes|Genetics. 12, jkac081 (2022).